課題1 細胞内分子ダイナミクスのシミュレーション
細胞内分子ダイナミクスのシミュレーション

代表
杉田 有治
理化学研究所
核内混み合い環境でのヌクレオソーム、クロマチンの機能発現機構
概要
分子シミュレーションの計算手法の開発と応用研究に取り組み、これまで到達することができなかった細胞スケールの生命現象を解明し、将来的な医療・創薬のデザインの実現を目指します。
細胞の中の多くは水ですが、タンパク質なども30%以上を占め、ひしめきあっています。細胞環境下でのタンパク質の長時間の振る舞いを計算し、細胞機能との関係を明らかにしていきます。
病気の多くはタンパク質の機能不全ともいえる現象がきっかけで起こります。そのため、最近の実験の多くは細胞内でのタンパク質の振る舞いをあるがままに研究しようとしています。一方、これまでの小さな計算機では到底そのような条件での計算はできません。単純化し、溶液の中で孤立した1つのタンパク質の計算をせざるをえませんでした。私たちは、スーパーコンピュータ「京」の優れた計算能力によって、これまでにないスケールで細胞環境に近い条件でタンパク質の働く姿をとらえたいと考えています。
【下動画】
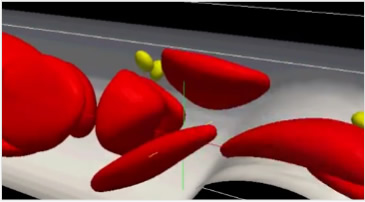
二重鎖DNAは、遺伝情報の担い手ですが、細胞の核の中では、コンパクトにまとまっており、クロマチンという構造を形成しています。遺伝子が働くためにRNAに転写されるときは、クロマチン構造が緩む必要があります。この構造変化はヒストンの化学修飾などによって制御されています。このような複雑な生命のしくみを研究するために、スーパーコンピュータ「京」の中に、クロマチンを構成するヌクレオソームの立体構造をバーチャルに構築して、運動方程式などの物理法則に基づいた分子動力学シミュレーションを実行することで、クロマチン構造がダイナミックに動く様子をシミュレーションして観察することができるようになりました。
概要
多くの医薬品はタンパク質を標的としており、より効果的な薬を見つけ出すためには、生体内の標的タンパク質と強く相互作用する化合物(リガンド)を探し出す必要があります。私たちは、スーパーコンピュータ「京」を用いた分子動力学計算により、標的タンパク質と薬の候補である化合物を含む系のシミュレーションを行っています。タンパク質と化合物間の結合の強さを精密にそして高速に計算し、標的タンパク質だけに強く作用する新しい化合物を設計することにより、短時間で効率よく低分子医薬品を開発することをめざしています。
5年間の取り組みと成果 BioSupercomputing Newsletter Vol.14より
論文
1. Doki S, Kato HE, Solcan N, Iwaki M, Koyama M, Hattori M, Iwase N, Tsukazaki T, Sugita Y, Kandori H, Newstead S, Ishitani R, Nureki O:
“Structural basis for dynamic mechanism of proton-coupled symport by the peptide transporter POT“
Proc Natl Acad Sci U S A, 110 11343-8 (2013)
2. Jaewoon Jung, Takaharu Mori, Yuji Sugita:
“Efficient lookup table using a linear function of inverse distance squared“
J. Comput. Chem., 34, 2412-2420 (2013)
3. Michael Feig, Yuji Sugita:
“Reaching new levels of realism in modeling biological macromolecules in cellular environments“
J. Mol. Graph. Model., 45, 144-156 (2013)
4. Jaewoon Jung, Takaharu Mori, and Yuji Sugita:
“Midpoint cell method for hybrid (MPI+OpenMP) parallelization of molecular dynamics simulations“
J. Comput. Chem., 35, 1064-1072 (2014)
5. Kono H, Shirayama K, Arimura Y, Tachiwana H, Kurumizaka H:
“Two arginine residues suppress the flexibility of nucleosomal DNA in the canonical nucleosome core“
PLoS ONE.10:e0120635. (2015)
6. Naoyuki Miyashita, Suyong Re, Yuji Sugita:
“REIN: Replica-exchange INterface for simulating protein dynamics and function“
International Journal of Quantum Chemistry, 115, 325-332 (2015)
7. Michael Feig, Ryuhei Harada, Takaharu Mori, Isseki Yu, Koichi Takahashi, Yuji Sugita):
“Complete atomistic model of a bacterial cytoplasm for integrating physics, biochemistry, and systems biology“
Journal of Molecular Graphics and Modelling, 58, 1–9 (2015)
8. Yuki Shindo, Kazunari Iwamoto, Kazunari Mouri, Kayo Hibino, Masaru Tomita, Hidetaka Kosako, Yasushi Sako & Koichi Takahashi:
“Conversion of graded phosphorylation into switch-like nuclear translocation via autoregulatory mechanisms in ERK signalling”
Nature Communications 7, Article number:10485, (2015)
体制図
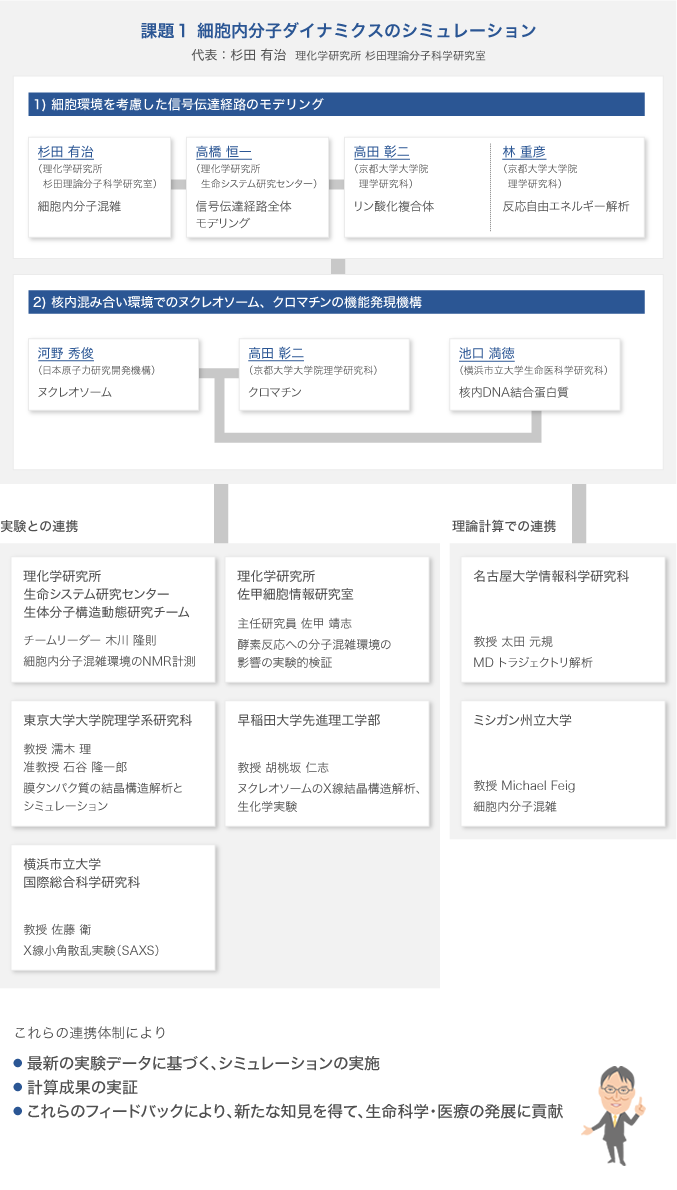
※2015年4月1日現在