課題1 細胞内分子ダイナミクスのシミュレーション
平成25年度~27年度における実施計画
研究開発課題の概要
蛋白質や核酸、脂質二重膜などの分子動力学計算は、現在でも盛んに行われている。しかし、計算機の演算性能の限界により、溶液中や脂質二重膜中での単一蛋白質の計算を行っているにすぎない。生命現象の理解、予測を実現していくには、分子レベルの計算とシステムズバイオロジーを融合し、「細胞まるごと」を理解することが必要不可欠である。本研究課題は「細胞まるごとシミュレーション」に至る必要なステップとして「細胞環境を強く意識した分子および細胞スケールシミュレーション(細胞内分子ダイナミクス)」を行うことを提案する。この研究の特徴は、①「京」の数十万CPUcoreを利用した大規模シミュレーションを実施すること、②細胞内のタンパク質の運動を観て、計測データと直接比較することで新しい生命現象の理解と予測を行うこと、③細胞内では蛋白質や核酸が混雑した環境である事を意識して、細胞質内と核内のダイナミクスと機能の解明に焦点を充てること、である。
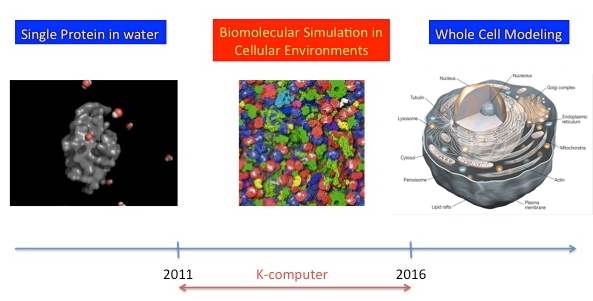
図. 分子レベルの計算から「細胞まるごと」の理解への過程で、「細胞内分子ダイナミクス」の理解と予測が必要である
図(中央) McGuffee SR and Elcock AH (2010) PLoS Computational Biology (2010) 6(3):e1000694
図(右) Copyright @ 2012 Life Technologies Corporation. Used Under permission.
「細胞内分子ダイナミクス」の理解と予測を実現するために、当初、以下の3つのサブ課題を実施し、研究プロジェクトの終盤ではこれらの知見を生かして「細胞まるごとシミュレーション」へ至る研究課題を明確にすることを目的とした。①細胞環境下での信号伝達経路のモデリング(高橋(理研) 、杉田(理研) 、太田(名大) ) 、②膜タンパク質による細胞膜を隔てた物質輸送(杉田(理研) 、石谷(東大) 、池口(横浜市大:H25から) 、林(京大:H25から) 、③核内DNAタンパク質の動的構造と機能の解明(河野(原子力機構) 、高田(京大:H25から) ) 。①では高橋らによる一分子粒度シミュレーション(システムズバイオロジー)と太田・杉田らによる分子動力学計算を連携して新しい研究分野を開拓する。②では「京」の計算資源を最大限に利用した「長時間分子動力学」計算に基づく自由エネルギー計算を行うことで「定量性」あるいは「予測性」を持つ分子シミュレーションを達成する。膜タンパク質は今後、創薬ターゲットになりうるがそのダイナミクスを本質的に理解しない限り、阻害剤などの設計は困難である。③では核内DNAタンパク質の相互作用を全原子および粗視化分子動力学計算を用いて解析する。ヒストンタンパク質やDNAのアセチル化やメチル化など「エピジェネティクス」は老化や分化などにおいて重要な役割を果たしているが、その構造やダイナミクスの理解はこれからである。以上のサブ課題を行うために、年度毎にソフトウェアの高度化の進行状況と研究課題の緊急性を考慮して、重点化をはかる。
しかし、平成24年度末に課題の選択と集中を行うために、計画を変更し、上記①と③のみを行うことになった。これにともない、太田(名大)が杉田(理研)チームの研究協力者に、石谷(東大)、池口(横浜市大)、林(京大)らは研究内容を変更してそのまま分担者として課題に参加することになった。
この研究課題においては、最先端計測との連携が欠かせない。理研生命システム研究センター(QBiC) では、‘最先端計測’、‘高性能計算’、‘機能デザイン’を3つの柱として研究を進めている。本研究課題では、QBiCの最先端計測部門や、その他の国内のin Cell NMRやXFELなどの最先端計測と連携をはかり、「計算(理論)」と「実験(計測)」の両面から生命科学の発展を目指していく。また、石谷らは東大濡木研究室でX線結晶構造解析と分子動力学計算の両方を行っている。「京」を用いた大規模分子動力学計算を濡木研究室で解かれた最新の結晶構造に関して適用していくことで「構造とダイナミクス」両方の知見を一度に得ることができる。この手法が成功するならば、「機能解析や創薬デザイン」を行うための必要な方法として生命科学に強いインパクトを与えるものになるだろう。
研究開発課題の内容
「細胞環境を考慮した信号伝達経路のモデリング」
- 1) 信号伝達経路上のリン酸化酵素への細胞質分子混雑の影響(杉田有治・理化学研究所)
- 「細胞内分子ダイナミクス」研究では、これまでの水中、脂質二重膜中、真空中などの環境ではなく、細胞環境(細胞質、生体膜、核など) を露に考慮した環境での生体分子ダイナミクスを研究する。細胞質の環境については、近年in cell NMRなどの実験が進んでおり、分子混雑などによるタンパク質動態への影響が調べられつつある。また、生体膜に関してもラフトなどが機能発現の場として重要であることが明らかになりつつある。そのような細胞環境による影響を計算と実験の両面から解析していくことで細胞環境の理解を深めることができる。マルチスケール・マルチレゾリューション分子動力学計算法を使う上で、多分子のサンプリング効率を高める工夫を行うとともに、細胞内に含まれる複数のタンパク質を含む分子動力学計算を行うことで、「細胞内」に近い環境を計算機中で構築し、溶液中や脂質二重膜中のタンパク質動態と比較する。そのような解析に適したトラジェクトリ解析を行う必要があり、名大太田らと協力する。「細胞内環境」での分子ダイナミクス、水和、タンパク質・タンパク質相互作用の計算を行う中で、相互作用を分割して、これまで考慮されていなかったターゲットタンパク質近傍の(別の)タンパク質や脂質分子からの相互作用を検討する。これにより、一部の膜タンパク質で見られるような脂質分子特異性やIn vivoとIn vitro実験の違いなどを説明する。
- 2) 細胞内環境下での信号伝達経路のモデリング基盤整備およびコード開発(高橋恒一・理化学研究所)
- 本研究では、「細胞内分子ダイナミクスのシミュレーション」研究の一環として、「京」の計算能力を活用し、一分子粒度かつ細胞まるごとスケールで、細胞表面の受容体が信号分子を感知してから核内で転写因子が遺伝子の発現のスイッチを入れるまでの一連の反応の計算を行う事を目指す。細胞環境下における分子の運動や反応ダイナミクスの実験情報は完全ではないが、一分子追跡や蛍光相関分光法による計測では、一次近似としてブラウン拡散運動として説明出来る範囲が予想以上に広い事が明らかになっている。本研究では一分子粒度反応拡散系モデルを用いるが、計算結果を一分子観察実験と精密に比較対照することで細胞内環境における細胞骨格による空間の構造化や巨大分子混雑による異常拡散などの影響を含め、「京」以後のスパコンによるさらに精密な計算への道筋を付ける事も目標の一部である。計算には微視格子反応拡散法を実装したSpatiocyteソフトウェアを用いる。これは、分子を六方最密格子で離散化された空間の格子点として表現し、分子の移動と反応を離散イベントとして駆動する手法であり、E-Cellシミュレーションプラットフォーム上に実装されている。本課題では、時間スケール、空間スケール、および反応ネットワークの複雑さ、の3つの次元での規模の大きさが挑戦となる。時間および空間スケールに関しては、「京」の計算能力で乗り越えたい。残った反応ネットワークの複雑さに関しては従来の静的な反応ネットワーク記述から動的なルールベース記述に切り替える事で対処する。ルールベースでの反応ネットワークモデリング技術自体はロスアラモスのBioNetGenや米国分子科学研究所のMoleculizerなど既存のものがあり、これを参考に一分子粒度シミュレーションに適した手法を開発実装する。
課題4では「ゲノム情報」を用いることによって細胞内に存在する反応ネットワークを推定しているが、ここではその実態を一分子粒度という近似を用いて解析している。研究交流を通して、「ゲノム情報」と「一分子観察実験」の間を担い、新しい研究分野を構築することを目指す。 - 3) 信号伝達経路上のリン酸化酵素の反応性解析(林重彦・京都大学大学院理学研究科)
- 林らはQM/MM自由エネルギー計算法を開発しており、「京」を用いた大規模計算を行うことでリン酸化反応などの酵素反応の理解と予測に繋がる研究を推進する。特に、MAPK シグナル伝達経路を構成するキナーゼである MEK(MAPKK)や ERK(MAPK)のリン酸化反応とその制御機構を明らかにする。杉田らの分子動力学計算によって得られたスナップショットを利用して、キナーゼ単体、及び複合体におけるリン酸化反応性を解析し、更に、混みあい環境における反応性の影響を明らかにする。
「核内混み合い環境でのヌクレオソーム、クロマチンの機能発現機構」
- 4) 全原子モデルにもとづくヌクレオソームポジション変化の自由エネルギープロファイル計算 (河野秀俊・日本原子力研究開発機構)
- 本研究では、「細胞内分子ダイナミクスのシミュレーション」研究の一環として、核内DNAタンパク質複合体の構造予測と機能解明のための研究開発を実施する。核内DNAは、ヒストンタンパク質に約150塩基対のDNAがほぼ2回巻きついたヌクレオソーム構造を基本構造とし、このヌクレオソームがコンパクトに凝集した構造をとっている。近年、このヌクレオソーム構造のポジションやその変化が遺伝子制御に深く関わっていることが明らかになってきた。また、エピジェネティクスの観点からも、ヌクレオソームを構成するヒストンの化学修飾、ヒストン変異体及びDNAの化学修飾が重要な役割を果たしていることがわかってきた。現在、カノニカルヒストン、ヒストン変異体及び化学修飾されたヒストンを含むヌクレオソームなど少なくとも27種類の結晶構造が明らかにされている。本研究では、これらの構造をもとにシミュレーション計算を行い、化学修飾や変異体、巻きついたDNAの塩基配列の違いがヌクレオソームのポジション変化や構造安定性に与える影響を明らかにすることにより、遺伝子制御メカニズムの一端を明らかにすることを目指す。シミュレーション計算で示唆された重要なアミノ酸残基、化学修飾、DNA塩基配列等について、生化学的な検証実験や立体構造決定が行われるよう実験系研究グループ(早稲田大学、胡桃坂研究室など) との連携を図りながら研究を進める。
本研究では、これまでにおもに原子力研究機構で開発してきたソフトウェアSCUBAを用いて大規模全原子分子動力学計算を実施する。このソフトウェアは、高い並列化性能を持つだけでなく、本研究を進める上で必要不可欠な機能であるフレキシブルな拘束条件の導入がなされている。SCUBAをさらに「京」に最適化、並列化していくことでヌクレオソームポジション変化の自由エネルギープロファイルを効率的に描き、ウェット研究者に必要な情報を提示することができる。 - 5)核内DNA結合タンパク質の機能ダイナミクス研究(池口満徳・横浜市立大学)
- 本研究では、「細胞内分子ダイナミクスのシミュレーション」研究の一環として、核内DNA結合タンパク質の機能ダイナミクスの研究を実施する。本グループでは、これまで分子動力学シミュレーションと、X線溶液散乱やNMRなど溶液系の構造生物学データを組み合わせる方法を開発してきた。本研究では、この方法を、クロマチン関連因子やDNA修復タンパク質など核内DNA結合タンパク質複合体に対し適用することで、その機能ダイナミクスの解析を行う。この際には、実験系研究グループと連携をもちつつ研究を進める。本研究では、次世代生命体統合シミュレーション研究開発プロジェクトにて開発してきたソフトウェアMARBLEを用い、さらに「京」に最適化をして活用していく。
- 6) 遺伝子の転写調節に関わる因子の分子動力学シミュレーションによる制御機構の解明研究(石谷隆一郎・東京大学大学院理学研究科)
- 細胞内信号伝達経路上にある遺伝子の転写調節にかかわる因子,HHMやβカテニンについて分子動力学シミュレーションを行い,その調節メカニズム解明を目指す高田らは、「信号伝達」と「クロマチン」の両方の課題について研究を行う予定である。
- 7) 粗視化分子モデルを用いた信号伝達経路上のリン酸化酵素複合体とクロマチンの動的モデリング(高田彰二・京都大学大学院理学系研究科)
- 高田らは、粗視化分子モデルを用いた大規模分子シミュレーションによって、信号伝達におけるリン酸化酵素複合体MEK-ERKの動的モデリングを行うとともに、ヌクレオソーム多量体からなるクロマチンが核内混雑環境でどのような携帯で超高密に折れたたまれているのかを解析する。
具体的な成果目標
各サブ課題の5年後の成果目標としては、
①原子粒度分子シミュレーションと分子粒度細胞シミュレーションを段階的に接続する粗視化モデルを開発し、細胞環境のタンパク質ダイナミクス、安定性、水和、分子認識に与える影響を明らかにすること。すなわち、In cell NMRが実現したように、In vivoの環境で始めて実現する生命現象を計算機中でも再現し、分子生物学者に必要な情報を提供できること、
②ヌクレオソームを構成するヒストンタンパク質の化学修飾、ヒストン変異体やDNAの化学修飾による遺伝子制御を理解するために、自由エネルギー計算法による定量的な解析を行い、修飾や変異の影響を定量的に予測可能にすること。これにより構造とダイナミクスの新しい観点から、エピジェネティクスを理解、予測していくことである。
しかし、既に述べたようにこの課題を行う大きな目的は、現在行われている「単一タンパク質の分子動力学」から「細胞まるごとシミュレーション」に至る必要なステップとして「細胞内分子ダイナミクス」の理解と予測に挑むことである。従って、研究開発プロジェクトの後半には、「細胞まるごとシミュレーション」に至る道筋が少なくとも現在よりも明確に示されていなければならない。









