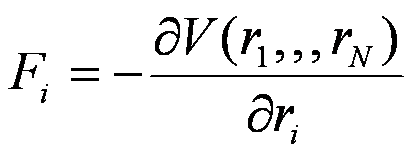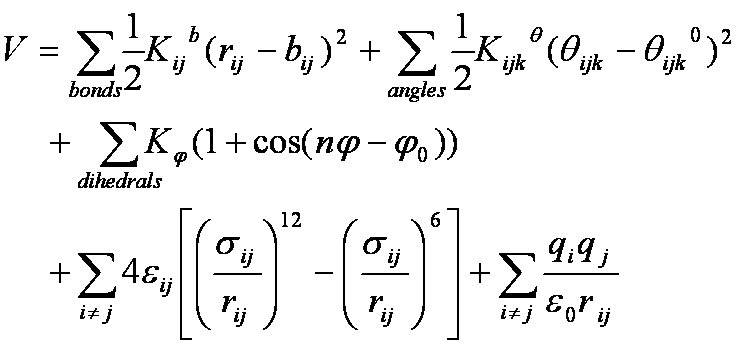GROMACS
1. プログラム名
GROMACS(以下、主な情報はこちらから得ています)
2. 内容
概要
極めて多様な目的に利用しうる分子動力学計算のパケージです。分子動力学計算ではタンパク質などの動的な挙動を解析しうるとともに、自由エネルギーなどの統計力学量をも求めることができます。
詳細
分子動力学計算はタンパク質など個々の分子を多くの原子の集まりとして考え、古典的なニュートンの運動方程式を解きます。
ここでriは原子iの位置ベクトル、Fiは原子iに作用する力、Nは原子の総数を示します。力Fiはポテンシャル関数Vから次式で求めることができます。
古典力学では本来的にそのポテンシャル関数を求めるための構成原理はなく、経験的に構成されます。そのため、個々の分子動力学計算ソフトウェアによってポテンシャル関数のの関数型やそこに用いられるパラメータは異なります。
GROMACSでは下記のポテンシャル関数が用いられています。詳細はGROMACSの最新のマニュアルを参照ください。
以下に、分子動力学計算を行うにあたっての留意点を示します。
1. 原子は「電荷と質量を持つ質点」とする。
2. ニュートンの運動方程式を解く。
3. ポテンシャル関数Vの構成原理はないため、ポテンシャル関数は経験的に決められる。
4. そこに含まれるパラメータは、分光学などの実験結果を踏まえ、あるいは量子論により、さらには実験結果と整合性が取れるように決められている。
5. それらパラメータを用いた分子動力学計算は、広範な実験結果を説明し、かつ新たな知見を生み出している。
6. ただし、「分子動力学計算が常に最適な選択」というわけではない。なお、ここでは述べませんが、GROMACSのパケージには粗視化モデル(例:1残基を1質点でモデル化)やブラウン動力学による解析方法も含まれている。
3. どんなことができるか
- 水溶液中のタンパク質の動的挙動を解析できます。
- タンパク質とリガンドの結合自由エネルギーなどの統計力学量を計算できます。
4. 関係論文
[1] Berendsen, et al. (1995) Comp. Phys. Comm. 91: 43-56.
[2] Lindahl, et al. (2001) J. Mol. Model. 7: 306-317.
[3] van der Spoel, et al. (2005) J. Comput. Chem. 26: 1701-1718.
[4] Hess, et al. (2008) J. Chem. Theory Comput. 4: 435-447.
[5] Pronk, et al. (2013) Bioinformatics 29 845-854
5. チュートリアル資料など
- 江口 至洋「SCLS計算機システムの実習-分子動力学の基礎-」
- 波内 良樹「GROMACS実習」
- 波内 良樹「GROMACSを使用した温度レプリカ交換分子動力学法(T-REMD)実習」
- 江口 至洋「温度レプリカ交換分子動力学法についてのQ&A」
6. 関連する教科書
- 柳田 敏雄他「計算と生命」岩波書店(2013)
- 岡崎 進、吉井 範行「コンピュータ・シミュレーションの基礎」化学同人(2011)
7. マニュアル
GROMACSの利用者マニュアルを参照ください。
8. 処理の手順
GROMACSを用いて様々なシミュレーションを行うことが可能です。一例として「5. チュートリアル資料など」にあるGROMACS実習では以下の手順のシミュレーションについて、SCLS計算機システムにおける実行例を紹介しています。
1. 解析対象となる分子の立体構造を得る (タンパク質の場合、通常はPDBから)
2. 欠失領域を埋める(含む、側鎖や水素原子)
3. SS結合の確認、ヒスチジンなどの電荷状態の確認など
4. GROMACSのトポロジーファイルを作る
5. 水分子を付加する(イオンを付加する)
6. 最急降下法などでエネルギーの最小化を行う
7. 平衡化シミュレーションを行う(例: NVT→NPTアンサンブル)
8. プロダクト・ランを行う
9. シミュレーション結果(trajectory data)を解析する
9. ソフトウェアのダウンロード
GROMACSは、こちらからダウンロードできます。
(1)スーパーコンピュータ「京」での実行
GROMACSについては、スーパーコンピュータ「京」での実行環境の構築手順をご案内しています。ご希望の方は、SCLS計算機システム![]() までご連絡ください。
までご連絡ください。
(2)SCLS計算機システムでの実行
GROMACSについては、SCLS計算機システムでの実行環境を整えています。利用ご希望の方は、SCLS計算機システム![]() までご連絡ください。
までご連絡ください。