EEM
1. プログラム名
EEM
2. 開発責任者
新井田 厚司(東京大学)
3. 主な開発者
新井田 厚司(東京大学)
4. 内容
概要
遺伝子セット情報に基づいてmRNA発現データ中で共発現している遺伝子群、発現モジュールを抽出します。
詳細
EEM法は遺伝子セット(Seed Gene Sets)情報に基づいてmRNA発現データ中で共発現している遺伝子群、発現モジュールを抽出するソフトウェアです。このEEM法の特徴は生物学的に解釈しやすい結果を得られるところにあり、実際EEM法を複数のがんの発現データに適用し、がんの転写プログラムを明らかにしています。
遺伝子セットは、個々の遺伝子のcisエレメントの解析から「同一の発現制御を受けている」と推定される遺伝子セットや、Chip-chip解析から「同一の発現制御を受けている」と推定される遺伝子セット、さらには個々の遺伝子のlocus情報から同じく「同一の発現制御を受けている」と推定される遺伝子セットなどが考慮されています。
遺伝子セットの情報と発現データから発現モジュールを抽出する過程を図1に示します(関係論文[1]を参照ください)。
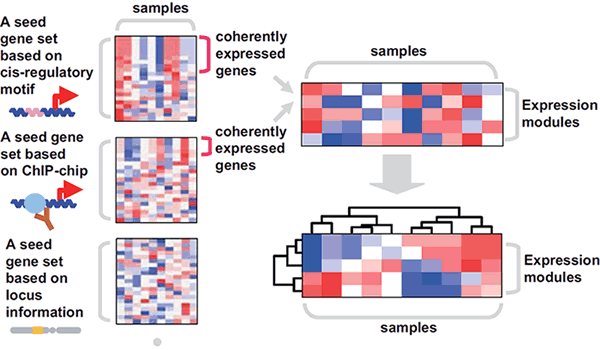
図1 Schema of systematic search for expression modules.
We prepared a collection of seed gene sets based on cis-regulatory motifs, ChIP-chip and gene locus information. We next statistically evaluated whether each seed gene set includes a significant large number of coherently expressed genes in expression profile data. If such a coherently expressed gene subset exists, we assumed it as an expression module, and obtained its averaged expression profile as an activity profile.
From Niida et al. BMC Bioinformatics 2009 10:71 doi:10.1186/1471-2105-10-71
関係論文[1]では乳がんの発現データから10の発現モジュールを抽出しています。発現データのサンプルからトリプルネガティブの乳がんのサンプルに注目すると、発現モジュールのうちE2F発現モジュール(34遺伝子から構成されている)とNFY発現モジュール(27遺伝子から構成されている)に含まれる遺伝子はupregulateされており、ER発現モジュール(17遺伝子から構成されている)はdownregulateされているとの結果が得られており、EEM法が発現データから意味のある情報を抽出する能力の高さを示しています。
5. どんなことができるか
- mRNA発現データから、生物学的に解釈しやすい発現モジュール(遺伝子集合)を抽出できます。
6. 関係論文
[1] Niida A et al.(2009) Gene set-based module discovery in the breast cancer transcriptome, BMC Bioinformatics. 26;10:71.
7. チュートリアル資料など
8. 関連する教科書
[1] 藤渕 航,堀本勝久(編)「マイクロアレイデータ統計解析プロトコール」(2008)羊土社
9. マニュアル
EEMの利用者マニュアルを参照ください。
10. 処理の手順
(1)ソースコードのコンパイル
実行環境に合わせてソースコードをコンパイルします。コンパイル環境の設定サンプルとして、GNUコンパイラ用、Intelコンパイラ用、富士通コンパイラ用を用意しています。
(2)コンパイルしたプログラムの実行
コンパイルしたプログラムを実行します。実行例として、入力用のサンプルデータと実行結果を用意しています。
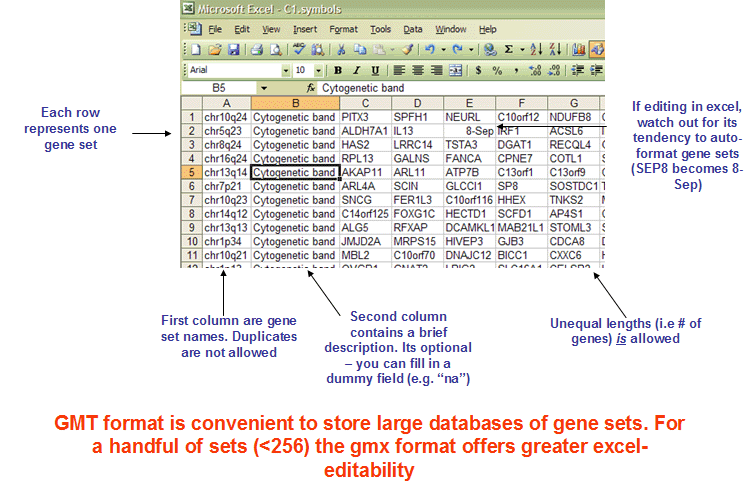
注1)入力データである遺伝子セット(Seed Gene Sets)は下記GMT形式です。
The GMT file format is a tab delimited file format that describes gene sets. In the GMT format, each row represents a gene set; in the GMX format, each column represents a gene set.
注2)入力データである発現プロファイルデータは表頭に実験名、表側に遺伝子名が記されています。EEMのプログラムの中で自動的に、各行を平均0分散1に標準化します。
注3)出力される「center=」は論文[1]にあるactivity profileを示しています。
注4)出力されるp-valueは-log10scaleでしめしています。出力の数字がaの場合p-valueは10-aとなります。
11. ソフトウェアのダウンロード
EEMは、ここからダウンロードできます。
(1)SCLS計算機システムでの実行
ソースコードのコンパイルは、富士通コンパイラ用設定サンプルを使用します。
12. 利用許諾権(ライセンス)について
EEM is Free Software, available under the GNU Lesser General Public License.









