生命の営みを突き詰めると生体内での原子・分子の反応や動的振る舞いに帰着するが、その運動は量子力学や統計熱力学などの物理法則に支配されている。水分子、低分子化合物、タンパク質、核酸などの生体分子の常温での物理的相互作用は分子力場で上手く記述されるが、我々は先進的な量子分子軌道計算を駆使して、これらの分子を統一的に扱う高精度なFUJI 分子力場を開発した。数値シミュレーションの結果は実験データと定量的に比較検証される必要があるが、タンパク質と薬候補化合物との結合自由エネルギーを非平衡統計力学のJarzynski等式を用いて導出するMP-CAFEE 法を開発して、薬開発の中で頻繁に測定される解離定数との定量的比較を可能にした。この方法で使用する分子動力学プログラムGROMACS の開発者のストックホルム大学Lindahl 教授と「京」に最適化したSIMD 計算カーネル※を開発してコンパイラだけで最適化した場合の二倍高速な計算を実現した。標的タンパク質は大学、IT 企業、製薬企業の三者で検討して決めて、フラグメント法による新規化合物の設計はIT 企業が、「京」による結合自由エネルギー計算を大学が、計算で活性が予測された化合物の合成とウェット実験は製薬企業が行なった。プロジェクトで取り上げたがん治療の為の標的タンパク質に対して薬としての必要条件を満した凡そ300 個の化合物に対して結合自由エネルギー計算を行った。この中から新薬として可能性がある化合物に対して動物実験が現在進行中である。

※ベクトル処理機能を用いてアセンブラレベルで高速化した分子動力学計算プログラム
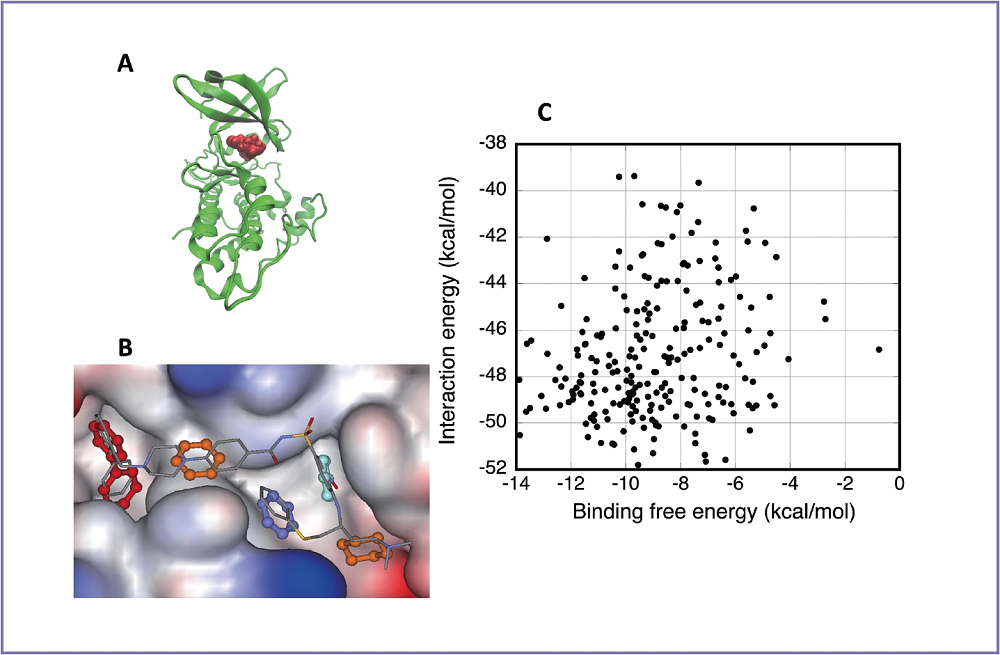
図:癌標的タンパク質と薬(A)、フラグメント法によるde novo 設計(B)、「京」でのMP-CAFEE計算(C)
Open Up Special Talk 4課題の5年間の取り組みと成果
課題1 細胞内分子ダイナミクスのシミュレーション
課題2 創薬応用シミュレーション
課題3 予測医療に向けた階層統合シミュレーション
課題4 大規模生命データ解析


















