「細胞内は約7割の空間を水が占めています」と聞くと、空間的に余裕のある環境を想像すると思いますが、実際はそうではありません。水以外の分子の占有率が30%の空間というのは、生体分子が非常に混み合っていて、たくさんのタンパク質や低分子、イオンなどが満員電車のようにごった返しています。そうした混雑環境でタンパク質や低分子がどのような動きや相互作用を示しているかは、生物学的にも、また創薬分野においても重要な問いです。しかし、細胞内の分子の動きは実験的測定が難しく、よく分かっていません。
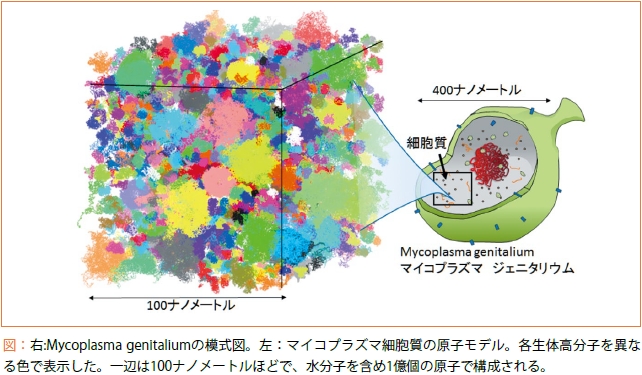
私たちは、スーパーコンピュータ「京」を用いた大規模な分子動力学(MD)計算(原子に働く力を計算し、運動方程式を繰り返し解くことによって、分子全体の動きをつぶさに追跡するシミュレーション方法)によって、細胞内における生体分子のふるまいを原子レベルで解明することを目指しています。具体的には、Mycoplasma genitaliumというバクテリアの細胞質をシミュレーションしています。
細胞質モデルは1000万原子~1億原子で構成されます(下図)。この超大規模モデルは、理化学研究所の研究員(森貴治研究員、原田隆平研究員)と、アメリカの共同研究者(Michael Feigミシガン州立大学教授)が作成しました[1]。マイコプラズマの遺伝子は全部分かっているので、タンパク質の種類がほぼ決定できます。また、生体高分子や代謝物分子の網羅的解析データがありますので、どんな分子がどれぐらいの濃度で存在するかも評価できます。このようにさまざまな実験データを用いることで、細胞膜とDNA以外の生体分子とその濃度がほぼ完全に「原子レベルで」再現できました。私は、こうしてできた「細胞質の丸ごとモデル」を、計算科学研究機構の研究員が開発したMDシミュレーションプログラム「GENESIS」[2]と「京」を使って動かしています。GENESISは、超大規模並列MDシミュレーションを可能にするためにさまざまな新技術が実装されたプログラムで、数十万個のCPUを同時に稼働させても効率が落ちません[3]。言い換えれば、私たちの研究は「京」の計算パワーとGENESISの高効率性が組み合わさって初めて成立するものです。
現在、シミュレーション自体はほぼ終了し、100ナノ秒程度の時間内に生体分子が細胞内でどのような動きをしているかを、いよいよ解析している最中です。タンパク質の拡散する速さなどは、実験値と良く対応していることが確認できました。その他にも細胞内の酵素の立体構造や代謝物の動きなどにおいて、試験管内の希薄な溶液環境では見られない多くの発見がありました。今後は創薬分野への応用も視野に入れて、細胞内におけるタンパク質間相互作用や代謝物と酵素の結合などを調査するために、より長時間のシミュレーションを実行する予定です。