血栓形成の初期過程における血小板凝集の
マルチスケールシミュレーション

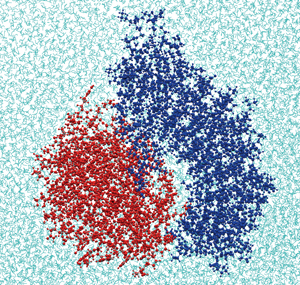
図1:血小板膜タンパク質GPIbαのN末端(青)とVWF A1ドメイン(赤)結合部の構造。
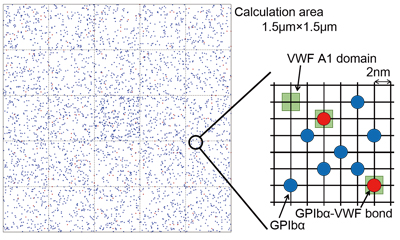
図2:血小板表面格子モデル。赤い点は結合しているGPIbα、青い点は結合していないGPIbαで血小板表面を移動する。
ケガなどによって血管が損傷した際、血管の損傷部位に血小板が凝集して、止血を行います。動脈硬化などによって血管の内壁が損傷した際にも血小板は凝集し、血管内に血栓を形成します。この血栓によって引き起こされる血栓症、即ち心筋梗塞や脳梗塞は、日本人の死因の4分の1を占める重要な循環器系疾患です。血小板は、その表面に存在するタンパク質glycoprotein Ibα (GPIbα)と血管内壁の損傷部に付着しているタンパク質von Willebrand因子(VWF)との相互作用によって血管内に凝集します。この2つのタンパク質の結合は血小板の接着面に数十個程度形成され、血小板を接着しています。また、より大きなスケールで見ると、血小板の接着には、血液中の赤血球の力学的作用が重要な役割を果たしています。
図1に示しているのは、分子動力学法によって得られたGPIbαとVWFの結合部の構造です。分子動力学法とは、タンパク質分子および水分子を構成する全原子について、ニュートンの運動方程式を解くことによって、各原子の時々刻々の位置を決定するという方法です。この方法を用いることによって、タンパク質間に働く力、および計算系のポテンシャルエネルギーを求めることができ、一対のタンパク質結合について、その結合形成、切断の反応速度をモデル化することが可能となります。図2に血小板表面の格子モデルを示します。ここでは血小板表面の膜タンパク質の挙動について、モンテカルロ法と呼ばれる統計的な手法を用いて解析を行い、時々刻々のタンパク質結合数および接着力を求めています。血小板表面積の約5分の1に相当する一辺1.5μmの計算領域中には4,000個のGPIbαが存在し、3,600カ所のVWFとの結合サイトが配置されています。図2の各ドットが1つのGPIbα分子を表しています。GPIbαは血小板表面を拡散しているとし、VWFは血管壁に固定されており、VWFと結合を形成しているGPIbαについては、拡散せず、膜上を移動しないとしました。また、VWF近くのGPIbαはある結合速度でVWFとの結合を形成し、結合はある切断速度で切断されるとしました。なお、これらの速度としては、分子動力学法からモデル化したモデルを使用することが可能です。ここで求めたタンパク質結合の数および接着力を有限差分法によるオイラー型流体-超弾性体連成手法に導入することで、赤血球の存在下で血小板が血管壁に吸着する現象を再現することが可能となります[1]。
このように、タンパク質スケールから血流スケールまでを繋ぐマルチスケールシミュレーションを行い、血栓形成のメカニズムの解明や、将来的には抗血小板薬の創薬、予測・個別化医療に役立つモデルの構築をめざして研究を進めています。
参考文献:
[1] BioSupercomputing Newsletter, Vol. 7, p. 7.