
大規模並列用MDコアプログラムの開発

理化学研究所 次世代計算科学研究開発プログラム
大野 洋介
(開発・高度化T)
我々のチームでは、「京」用に高速化されたコアプログラム・ライブラリの提供および「京」でのアプリケーション高速化技術の習得・蓄積を目的として、大規模並列用MDコアプログラムを開発してきました。
分子動力学(Molecular Dynamics, MD)シミュレーションは分子の運動・構造変化等を計算で求める手法で、生命科学においては、タンパク質等の生命現象の基盤となる生体分子を計算対象としています。シミュレーションでは原子間に働く力を計算し運動方程式を数値積分することで全ての原子の運動を計算します。運動方程式の数値積分と古典力場で近似した共有結合による力の計算は原子の個数に比例した計算量ですが、ファンデルワールス力(分子間力)とクーロン力は原子の組み合わせの個数に比例、つまり、原子の個数の二乗に比例した計算量が必要です。ファンデルワールス力は1.4nm以遠では非常に小さいので距離の離れた原子のペアの計算は省略するカットオフ法を使いますが、クーロン力は距離の二乗に反比例するので単純に無視することはできません。このため、FFTを使うことで遠距離クーロン力の計算量をO (N log N ) に抑えるPME(Particle Mesh Ewald)法が主流の計算手法となっています。しかし、FFTは並列化した場合の通信が多く大規模並列では効率が低下するため、大規模並列ではFMM(高速多重極展開法)等のFFTよりも通信の少ない手法の採用が進みつつあります。また、PME法は周期境界条件で厳密に正しい手法で、現実の細胞内のような周期性のない対象では擬似的な周期性が問題になる場合もあります。そのため、電荷・双極子モーメントが中性等の条件下で補正したカットオフ法が使われることもあります[1,2]。カットオフ法は通信はほぼ近接ノード間なので大規模並列にも適しています。
いずれの手法においても、計算時間の多くを何らかの形のカットオフ法計算が占めているので、本研究ではカットオフ法の「京」での最適化を重点的に行なってきました。最もよい条件ではカットオフ法計算部分の単体CPUでの計算効率は60%に達しました。2012年10月に行なった「京」全ノード評価(注1)では、5.4億原子・カットオフ距離2.8nm・ポテンシャル計算4ステップに1回の計算で実効計算速度4PFlops・効率38%を達成しました。ポテンシャルを毎回計算する場合では、79872ノードでの5.2億原子の計算で実効計算速度4.4PFlops・効率43%になりました。
並列性能をまとめたのが図1です。ノードあたりの計算規模(原子数)を一定にした場合の並列性(weak scaling)が緑の破線で、上から順にノードあたりの原子数が52338、6542、818、102の場合です。いずれの場合もほとんど一定の計算時間になっており、高い並列性が達成できています。全体の計算規模を固定した場合の並列性(strongs caling)が赤い線で、左から順に418707、3349656、7536726、26797248、214377984原子の場合です。並列性はノードあたりの原子数に依存しており、6000原子以上ではほぼノード数比例で性能が向上しましたが、ノード数を8、64倍にしノードあたり800、100原子になると、並列効率が50%、20%に低下しており、ノードあたり100原子程度で1ステップの計算時間10ミリ秒程度が実用上の限界だと考えられます。FMMによる遠距離クーロン力計算も実装中の試験評価ですが、カットオフ計算と比べて並列性の低下は少なく、「京」全ノード規模でも効率の低下は少ないことを確認しました。
本研究で得られた「京」での最適化の知見は他チームのアプリケーションの開発・最適化支援にもいかされています。また、このMDコアプログラムは、「京」での最適化の実例・参照コードとして、あるいは最適化済みの再利用可能MDコードとして、公開される予定です。
本研究は高度化チーム小山洋*、舛本現、長谷川亜樹、森本元太郎、沖本憲明、平野秀典との共同研究です。
* 現職 独立行政法人 物質・材料研究機構 (NIMS)
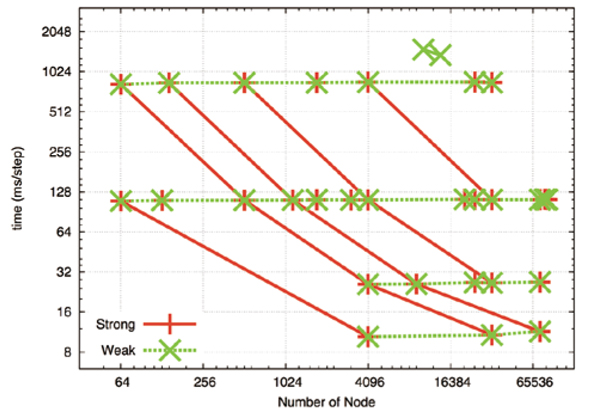
図1:カットオフ法の並列性能。カットオフ距離2.8nm。横軸はノード数、縦軸は1ステップの計算時間(ミリ秒)。緑破線がノードあたりの原子数固定(weakscaling)、赤線が全原子数固定(strong scaling)
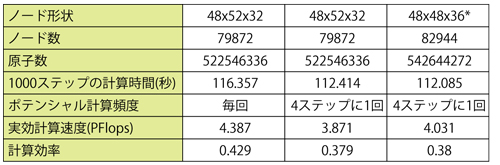
表1:カットオフ法の「京」全ノード規模計算性能。カットオフ距離2.8nm
表注*「京」のネットワーク形状48x54x32とは異なりますが今回は大きな影響はありませんでした。
注1
HPCIシステム利用研究課題として理化学研究所のスーパーコンピュータ「京」を利用(課題番号:hp120068)
【参考文献】
[1] Wolf, D. and Keblinski, P. and Phillpot, SR and Eggebrecht, J., J. Chem. Phys. 110 8254 (1999)
[2] Fukuda, I. and Yonezawa, Y. and Nakamura, H., J. Chem. Phys. 134, 164107 (2011)
BioSupercomputing Newsletter Vol.8
- SPECIAL INTERVIEW
- 革新的なアプローチでライフサイエンス分野の未来を切り拓いてきたグランドチャレンジ
理化学研究所 次世代計算科学研究開発プログラム
プログラムディレクター 茅 幸二 - ライフサイエンス分野の研究開発に革新をもたらした画期的なプロジェクト
理化学研究所 次世代計算科学研究開発プログラム
副プログラムディレクター 姫野 龍太郎
- 研究報告
- マルチスケール・マルチフィジックス心臓シミュレータUT-Heart
東京大学新領域創成科学研究科
久田 俊明、杉浦 清了、鷲尾 巧、岡田 純一、高橋 彰仁
(臓器全身ケールWG) - 膵臓β細胞内インスリン顆粒動態シミュレーション・モデル
神戸大学大学院システム情報学研究科 玉置 久(細胞スケールWG) - 京による全脳シミュレーションへの道のり
理化学研究所 脳科学総合研究センター ユーリッヒ研究センター
神経科学・医療研究院(INM-6)
アーヘン工科大学医学部 マーカス・ディースマン(脳神経系WG) - 大規模並列用MDコアプログラムの開発
理化学研究所 次世代計算科学研究開発プログラム 大野 洋介(開発・高度化T)
- SPECIAL INTERVIEW
- 循環器系および筋骨格系・脳神経系における階層統合シミュレーションの実現をめざす
東京大学大学院工学系研究科機械工学専攻 教授 高木 周(課題3 代表) - 最先端シークエンサーによる大規模データを「京」で解析し生命プログラムとその多様性の理解を進める
東京大学医科学研究所ヒトゲノム解析センター 教授 宮野 悟(課題4代表)
- 報告
- 「4th Biosupercomputing Symposium」の開催報告
理化学研究所 次世代計算科学研究開発プログラム 田村 栄悦 - 京互換機:SCLS計算機システムの導入
理化学研究所 HPCI計算生命科学推進プログラム チーム員 木戸 善之
