
スーパーコンピュータを用いた大規模遺伝子
ネットワーク推定ソフトウェア SiGN

東京大学大学院情報理工学系研究科
玉田 嘉紀
(データ解析融合WG)
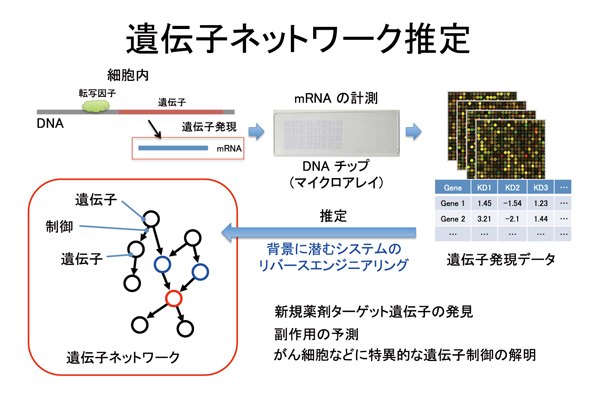
ヒトの細胞にはおよそ2万から3万の遺伝子があるといわれています。ヒトの体はそのほとんどがタンパクでできていて、遺伝子は細胞内でどのようなタンパクを作るのかという設計図に当たります。どのタンパクをいつどのくらい作ればいいかという制御も専用の遺伝子が行っており、その遺伝子(≒タンパク)もまた別の遺伝子によって制御されています。つまり遺伝子同士が複雑な制御のネットワークを形成しています。このネットワークはまだほとんどが解明されていません。同じヒトの細胞でも臓器によってネットワークは違います。また薬によっても変化し、がん細胞ではネットワークが壊れてしまっています。このような遺伝子の制御のネットワーク(=遺伝子ネットワーク)を計測可能なデータから数学的・統計学的・情報科学的な方法によって予測・推定しようというのが遺伝子ネットワーク推定です。現在の技術では細胞内で生成されているタンパク全てを計測することはできませんが、タンパクが生成される前段階で合成されるmRNAならば全ての遺伝子についてその量を計測することができます。このように計測したデータを遺伝子発現データといいます。1回の計測で得られるデータは、細胞のある状態の一瞬を捕らえたスナップショットです。この1回の計測データからでは遺伝子間の制御を予測・推定することは不可能で大量のデータが必要です。従って細胞に様々な刺激を与えたり、特定の病気の患者の細胞を集めたり、あるいは一定時間ごとに時系列にデータを計測したり、といったことで推定に必要なデータを集めます。遺伝子ネットワークを予測・推定することにより、これまで時間を掛けて一つ一つ遺伝子を探し実験を繰り返してきた遺伝子間の制御関係の解明を、計算によって網羅的に行うことで、新しい薬の開発、がん特異的な遺伝子の同定やその機能の解明が効率良くできるようになることが期待されています。
SiGN(サイン)は遺伝子発現データからスーパーコンピュータを用いて遺伝子ネットワークを推定するソフトウェアです。遺伝子ネットワークとして様々なモデルが提案されていますが、それぞれ一長一短があり最良のものというのはありません。またモデルが決まってもデータからそのパラメータを推定する方法にも複数あり、これまたどれも一長一短です。SiGNではスーパーコンピュータでの計算を前提とした膨大な計算時間の必要な複数の遺伝子ネットワークモデルと複数の推定アルゴリズムを実装した遺伝子ネットワーク推定ソフトウェアになっています。具体的には、SiGNは、静的・動的ベイジアンネットワークを用いたSiGN-BN、状態空間モデル (State Space Model: SSM) を用いたSiGN-SSM、L1正則化法によるパラメータ推定法を実装したSiGN-L1の3つのサブプログラムから構成されています。SiGN-BNはNNSR法という新しいアルゴリズムを搭載しており、これまで1000遺伝子程度が限界だったベイジアンネットワークを用いた遺伝子ネットワーク推定を全ゲノム(全遺伝子)に対しておこなう事ができるようになっています。SiGN-SSMは時系列データからシミュレーション可能な動的遺伝子ネットワークを推定しますが、ネットワークの構造自体は得られず全遺伝子間の関係の強度が数値で得られます。スーパーコンピュータによってこれまで難しかったネットワーク構造の計算が信頼度付きで計算可能になりました。L1正則化は元々大規模な遺伝子ネットワークに適用可能な方法ですが、個人の遺伝子発現の違いを考慮したネットワークを推定しようとすると、従来の方法では計算時間が足りませんでした。「京」を用いることによりこれも現実的な時間で計算することが可能になりました。
SiGNは「京」とヒトゲノム解析センターのスーパーコンピュータShirokane を主なターゲットとして開発していて、Shirokane ではすでにいくつかのサブプログラムがインストールされユーザが自由に使える状態になっています。詳細はSiGNのウェブサイトhttp://sign.hgc.jpまでどうぞ。
 |
BioSupercomputing Newsletter Vol.7
- SPECIAL INTERVIEW
- 「京」開発担当者に聞く今後のスパコン戦略とエクサスケールに向けた取り組み
富士通株式会社 テクニカルコンピューティングソリューション事業本部
エグゼクティブ・アーキテクト 奥田 基 - 「京」への展開で、実利用に向けた最適化とさらなる規模の拡張を進める大規模仮想ライブラリ
東京大学大学院工学系研究科 化学システム工学専攻 教授 船津 公人
- 研究報告
- 水の誘電率計算から得られる古くて新しい問題
大阪大学蛋白質研究所 中村 春木(分子スケールWG) - 大規模並列計算用流体・構造連成解析プログラムの開発
理化学研究所 情報基盤センター 杉山 和靖(臓器全身スケールWG) - スーパーコンピュータを用いた大規模遺伝子ネットワーク推定ソフトウェア SiGN
東京大学大学院情報理工学系研究科 玉田 嘉紀(データ解析融合WG) - ISLiM研究開発ソフトウェアのソース・コード公開に向けた活動
理化学研究所 次世代計算科学研究開発プログラム 田村 栄悦
- SPECIAL INTERVIEW
- 「京」を用いた大規模シミュレーションによって細胞内分子ダイナミクスの理解と予測を実現する
理化学研究所 基幹研究所 杉田理論分子科学研究室 主任研究員 杉田 有治(課題1 代表) - 日本の優れたコンピュータ技術を活かして革新的な分子動力学創薬に挑戦
東京大学 先端科学技術研究センター 特任教授 藤谷 秀章(課題2 代表)
- 報告
- 大学の新入生に行った計算生命科学の講義
理化学研究所 HPCI計算生命科学推進プログラム 鎌田 知佐
- 体制
- 計算科学技術推進体制
