
全原子モデルにもとづくヌクレオソームポジション変化の
自由エネルギープロファイル計算
HPCI戦略プログラム 分野1 予測する生命科学・医療および創薬基盤
日本原子力研究開発機構
量子ビーム応用研究部門
(左から) 河野 秀俊、石田 恒、米谷 佳晃、池部 仁善 (分野1-課題1)




ヒトの遺伝子は約30億塩基対からなるゲノムDNAにコードされ、その全長は約2mにもなります。ヒトを含めた真核生物のゲノムDNAは、ヒストンと呼ばれるタンパク質を芯にして、DNAが2回弱巻きついたヌクレオソーム構造を形成することで、直径約数μメートルの細胞核内にコンパクトに収納されています。ヌクレオソームはさらに、クロマチンと呼ばれる高次構造を形成しています。近年、それらの構造変化が遺伝子の発現制御において重要な役割を果たしていることがわかってきました。私たちの遺伝子はヌクレオソーム構造をとることによって、損傷から守られ安定に存在しています。一方、生命機能の根幹となる転写、複製、修復、組み換えといったDNAの代謝の際には、制御タンパク質やポリメラーゼと直接相互作用するために、ヌクレオソーム構造が壊されたり、その位置を変えたりすることが起こります (図1)。この一見すると相矛盾するような働きがヌクレオソーム構造にはあります。果たして、このようなヌクレオソームの構造破壊や位置変化(ポジション変化)はどのようなメカニズムで起こるのでしょうか。
さらに、ヌクレオソーム構造の変化は、細胞分化のメカニズムにも影響を与えることが分かってきました。私たちの体を構成する個々の細胞は、基本的に同じ遺伝情報(ゲノムDNA)を持っています。しかし、一度細胞が分化すると、一般には皮膚細胞からは皮膚細胞、肝細胞からは肝細胞しかできません。最近の研究により、そのからくりはヌクレオソーム構造の構成要素であるヒストンタンパク質の化学修飾と密接に関係することが明らかになってきました。このように、遺伝子の発現パターンがある種の記憶として維持、継承されていくことはエピジェネティクスと呼ばれ、盛んに研究されています。
本研究では、コンピュータシミュレーションを使って、ヌクレオソームのポジション変化を自由エネルギープロファイルの観点から明らかにしていきます。また、DNAの塩基配列の違い、ヒストンの化学修飾、変異体の影響が自由エネルギープロファイルにどのような形で現れるのか調べることによって、これらのエピジェネティクス研究を進めていきます。私たちはこれまで、SCUBAという大規模分子動力学シミュレーションに適したプログラムを開発してきました。SCUBAでは、対象の系を空間分割して並列に計算できるアルゴリズムをいち早く導入し、DNA相同組換え時に見られるHollidayJunction 分岐点移動や、200万原子を超える系でのリボソーム分子のダイナミクス解析を実行してきました。図2に示すように、ヌクレオソームは、熱揺らぎによって自発的にゆっくりと呼吸をするかのように揺らいだり(breathing)、ヒストンタンパク質に巻きついたDNAがほどけたり(opening)することが知られています。本課題では、そのようなヌクレオソームの運動メカニズムや構造安定性が、ヒストンの化学修飾、その変異体、さらに、巻きついたDNAの塩基配列の違いによってどのように変化するのかを、京コンピュータを使って明らかにしていきたいと考えています。このダイナミクスはサブ秒の時間オーダーであるため、世界最速である京コンピュータをもってしても、計算中に熱揺らぎの現象を待っているだけでは追いかけることはできません。そこで、適切な反応座標を設定し、その座標に沿って構造変化を誘導することによって、構造変化に対する自由エネルギープロファイルを計算します。このようなシミュレーション計算を通して、遺伝子発現がどのようにコントロールされているかという生命活動の基本的な分子メカニズムを、物理化学的に理解することを目指しています。
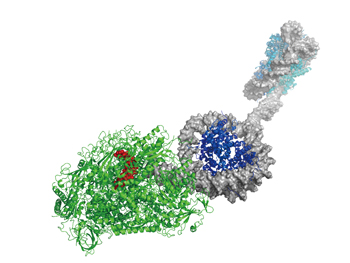 |
図1:RNAポリメラーゼによるmRNA合成。RNAポリメラーゼは、ヌクレオソーム構造をとっているゲノムDNAからどのように転写反応を進めているのでしょうか。そのような問題にシミュレーション計算を通して挑んでいます。 |
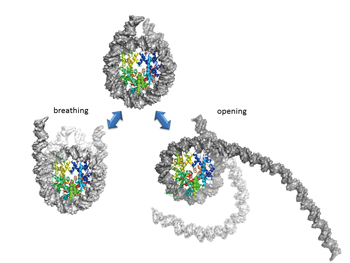 |
図2:シミュレーション計算によって、図に示したようなヌクレオソームの構造変化に対する自由エネルギープロファイル解析を行っています。ゲノムDNAは呼吸するかのようにヒストンタンパク質から離れたり巻きついたり、また、大きくほどけたりしていることが実験的に示唆されています。 |
BioSupercomputing Newsletter Vol.6
- SPECIAL INTERVIEW
- 新しい流体構造連成解析手法(ZZ-EFSI)の開発によっていち早く高い演算性能を達成
東京大学大学院工学系研究科 特任准教授 杉山 和靖 - 開発・高度化チームに聞く「京」の実力と高い性能を引き出すために続くチューニングの取り組み
理化学研究所 生命システム研究センター 生命モデリングコア計算分子設計研究グループ
グループディレクター 泰地 真弘人
理化学研究所 次世代計算科学研究開発プログラム 次世代生命体統合シミュレーション研究推進グループ
生命体基盤ソフトウェア開発・高度化チーム 上級研究員 大野 洋介
理化学研究所 HPCI計算生命科学推進プログラム 高度化推進グループ
高度化推進チーム 上級研究員 小山 洋
理化学研究所 次世代計算科学研究開発プログラム 次世代生命体統合シミュレーション研究推進グループ
生命体基盤ソフトウェア開発・高度化チーム 研究員 舛本 現
理化学研究所 次世代計算科学研究開発プログラム 次世代生命体統合シミュレーション研究推進グループ
生命体基盤ソフトウェア開発・高度化チーム リサーチアソシエイト 長谷川 亜樹
- 研究報告
- 全原子分子動力学シミュレーションによる多剤排出トランスポーターAcrBの機能解析
横浜市立大学大学院生命ナノシステム科学研究科 山根 努 / 池口 満徳( 分子スケールWG) - ヒト循環器系のマルチスケールモデリング
理化学研究所 次世代計算科学研究開発プログラム 梁 夫友( 臓器全身スケールWG) - 初期サッカード視覚運動系のスパイキングニューロンレベルでのモデル化
京都大学 ジャン・モーレン
奈良先端科学技術大学院大学 柴田 智広
沖縄科学技術大学院大学 銅谷 賢治
(脳神経系WG) - 大規模並列用MDコアプログラムの開発
理化学研究所 次世代計算科学研究開発プログラム 大野 洋介(開発・高度化T)
- SPECIAL INTERVIEW
- 複雑な生命現象の理解と予測に向けて計算生命科学の明日を拓く
理化学研究所 HPCI計算生命科学推進プログラム プログラムディレクター 柳田 敏雄
理化学研究所 HPCI計算生命科学推進プログラム 副プログラムディレクター 木寺 詔紀
理化学研究所 HPCI計算生命科学推進プログラム 副プログラムディレクター 江口 至洋
- 研究報告
- 全原子モデルにもとづくヌクレオソームポジション変化の自由エネルギープロファイル計算
日本原子力研究開発機構 量子ビーム応用研究部門
河野 秀俊 / 石田 恒 / 米谷 佳晃 / 池部 仁善
(分野1-課題1) - 骨格筋の活動の推定と脊髄反射の神経モデル
東京大学情報理工学系研究科 中村 仁彦(分野1-課題3)
- 報告
- ISLiM 成果報告会2011
理化学研究所 次世代計算科学研究開発プログラム 田村 栄悦 - 高等学校で行った計算生命科学の授業
理化学研究所 HPCI計算生命科学推進プログラム 鎌田 知佐 / 藤原 康広 / 江口 至洋
