
創薬応用シミュレーション
HPCI戦略プログラム 分野1 予測する生命科学・医療及び創薬基盤

東京大学先端科学技術研究センター
藤谷 秀章(分野1-課題2)
タンパク質とこれに特異的に結合する化合物(リガンド)の相互作用を調べる事は、生命現象の基礎的研究の大きな課題であると同時に応用の観点からも極めて重要である。特にタンパク質と薬分子との結合自由エネルギーを精確に求める事は創薬分野における永年の課題である。ドッキンング・シミュレーションを初めとして様々な方法が試みられて来たが、薬分子の設計に有効な精度でタンパク質との結合自由エネルギーを予測する方法はなかった。タンパク質とリガンドが水中(細胞液)で結合した状態と、それが別々に水中に溶けている二つの異なった熱平衡状態の間の自由エネルギー差を求めるには、タンパク質の構造変化や分子の運動によるエントロピー変化を精確に取り入れる方法が必要になる。この様な計算には分子動力学法が使われるがその精度は用いる力場パラメータに強く依存する。
タンパク質主鎖のラマチャンドラ角に対する捩れ相互作用はタンパク質立体構造を決める最も重要な力場パラメータであり、図1でこのエネルギー・プロファイルをグリシンペプチドで比較した[1]。黒い実線が今日最も精度の高い分子軌道計算(LCCSD)から求めたプロファイルで、AMBER(ff99,ff03)やOPLS-AAなど従来の分子力場と比較した。ff99sbは2006年に発表されたff99の主鎖の捩れ相互作用だけを修正した力場で、幾つかの角度で高精度分子軌道計算のエネルギーに一致する様に決められた。この為にφやψが0°でのエネルギー値は黒い線に近いが、φが80°から180°間では黒い線のエネルギーと比較して1kcal/mol以上高いエネルギー障壁が見られる。我々はAMBERの電荷とvander Waalsパラメータを使って、タンパク質やDNA・RNA、任意の有機化合物に対して統一的に力場パラメータを割当てる方法を開発してFUJI力場と名付けた[2]。
自由エネルギー差ΔGと非平衡仕事量Wの関係式を用いて[3]、リガンドの他分子に対する相互作用が完全に存在する状態から、相互作用が無くなるまでの複数の中間状態で分子動力学計算を独立に実行して[4]、隣の状態に移行する為に必要な仕事量Wから結合自由エネルギーを求める(図2)。このMP-CAFEE法とFUJI力場を用いてタンパク質とリガンドの結合自由エネルギーを正確に求める事が可能になる。
【参考文献】
[1] H. Fujitani, A. Matsuura, S. Sakai, H. Sato, and Y. Tanida : J. Chem. Theory Comput., 5, 1155 (2009).
[2] H. Fujitani, Y. Tanida, and Azuma Matsuura : Phys. Rev. E, 79, 021914 (2009).
[3] C. Jarzynski : Phys. Rev. Lett., 78, 2690-2693 (1997).
[4] H. Fujitani, Y. Tanida, M. Ito, G. Jayachandran, C. D. Snow, M. R. Shirts, E. J.Sorin, and V. S. Pande : J. Chem. Phys., 123, 084108 (2005).
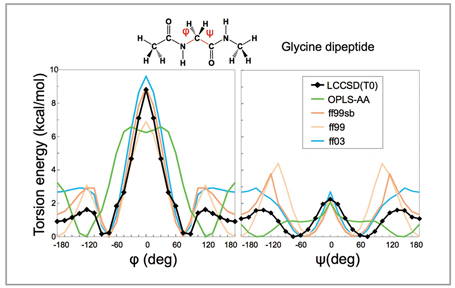 |
図1:タンパク質主鎖の捩れ相互作用の比較 |
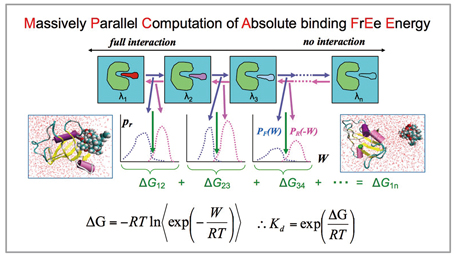 |
図2:超並列結合自由エネルギー計算法 MP-CAFEE |
BioSupercomputing Newsletter Vol.5
- SPECIAL INTERVIEW
- “予測する生物学”をめざすバイオスーパーコンピューティングの挑戦はいよいよ世界一の「京」で成果を出すフェーズに入った
理化学研究所 次世代計算科学研究開発プログラム 副プログラムディレクター 姫野 龍太郎 - 高性能計算機資源および開発アプリケーションの産業利用促進を図るために何をすべきか
計算科学振興財団 チーフコーディネーター 福田 正大
都市活力研究所 主席研究員 バイオグリッドセンター関西 理事・事務局長 志水 隆一
- 研究報告
- QM/MM 自由エネルギー法による酵素反応分子機構の解析
京都大学大学院理学研究科 林 重彦(分子スケールWG) - アクチン細胞骨格の計算メカノバイオロジー
京都大学再生医科学研究所 井上 康博(細胞スケールWG) - 血栓シミュレーションに向けた血流解析手法の開発
東京大学工学系研究科 伊井 仁志(臓器全身スケールWG) - 生命体シミュレーションのためのデータ同化技術の開発
統計数理研究所 樋口 知之(データ解析融合WG)
- SPECIAL INTERVIEW
- 複雑な生命現象の理解と予測に向けて計算生命科学の明日を拓く
理化学研究所 HPCI計算生命科学推進プログラム プログラムディレクター 柳田 敏雄
理化学研究所 HPCI計算生命科学推進プログラム 副プログラムディレクター 木寺 詔紀
理化学研究所 HPCI計算生命科学推進プログラム 副プログラムディレクター 江口 至洋
- 研究報告
- 創薬応用シミュレーション
東京大学先端科学技術研究センター 藤谷 秀章(分野1-課題2) - 次世代DNAシークエンサデータの超高速解析
東京工業大学大学院情報理工学研究科
秋山 泰 / 石田貴士 / 角田将典 / 鈴木脩司(分野1-課題4)
- 報告
- バイオスーパーコンピューティングサマースクール2011
理化学研究所 次世代計算科学研究開発プログラム
石峯 康浩(臓器全身スケールWG)
統計数理研究所 データ同化研究開発センター
斎藤 正也(データ解析融合WG)
新潟国際情報大学
近山 英輔(細胞スケールWG)
東海大学医学部内科学系循環器内科
七澤 洋平(細胞スケール/臓器全身スケールWG)
理化学研究所 次世代計算科学研究開発プログラム
半田 高史(脳神経系WG)
理化学研究所 次世代計算科学研究開発プログラム
舛本 現(開発・高度化T)
理化学研究所 次世代計算科学研究開発プログラム
森次 圭(分子スケールWG) - 「次世代生命体統合シミュレーションソフトウェアの開発(ISLiM)」開発アプリケーション紹介ページ、オープン
次世代計算科学研究開発プログラム 次世代生命体統合シミュレーション研究推進グループ
