次世代シークエンサーデータ解析のための情報処理システムの開発
DNAは生物の遺伝情報を担い、ヌクレオチドと呼ばれる単量体が鎖状に結合して構成されています。ヌクレオチドの並び(塩基配列)が判明すると、DNAに基づいて作成されるRNAや、そのRNAに基づいて作成されるタンパク質が推測できるようになります。DNA、RNA、タンパク質は、お互いに影響しながらさまざまな生体内の機能や化学反応に関与しているため、塩基配列を決定することで、生体物質間の複雑な相互作用を解明する端緒が開けます。また、生物種内や生物種間に類似した塩基配列があり、これらを比較することで類似部分の機能や進化について多くの知見を得ることができます。
DNAの塩基配列を自動的に読み取る装置であるDNAシークエンサーの発展により、塩基配列の読み取り速度とコスト当たりの読み取り量が急速に向上しています。約1日で1000億塩基対を読み取るものも販売されています。ヒトゲノムが約30億塩基対であることと比較すると、非常に高速であることが分かります。膨大なDNA情報を得ることが比較的容易になったため、現在はこれらの情報を高速に解析するシステムが求められています。そこで、私たちのチームでは、高速な解析アルゴリズムの開発と、複数の計算ノードを利用した並列計算により、膨大なDNA情報の解析を可能とする情報処理システムの開発を行っています。
近年、DNAシークエンサーの発展から、環境中(ヒトの体表・体内、土壌、水圏など)に存在する微生物集団のゲノム(メタゲノム)解析が盛んになっています。環境中に存在する微生物の組成(系統解析)や遺伝子の組成(機能解析)を環境間で比較することで、微生物とヒトの健康との関係や、環境と微生物集団の相互作用、微生物同士の相互作用について知見を得ようという試みです。環境中に存在する微生物や遺伝子を推定するために、読み取った塩基配列と類似した塩基配列を既知の塩基配列群から検索しますが、従来はこの計算に多くの時間が必要でした。
私たちは、検索アルゴリズムを改良することで、従来手法の100倍以上高速なGHOSTXを開発し、次いでGHOSTXを基に並列解析を行うGHOST-MPを開発しました。GHOSTXは、問い合わせ塩基配列と検索対象塩基配列群のそれぞれで接尾辞配列(Suffix Array)と呼ばれるデータ構造を構築し、索引として利用することで、全塩基配列を走査(逐次分析)することなく類似配列の探索を高速に行います。GHOST-MPは、複数の計算ノード間でデータの分配・収集と同時に検索を行うことで、検索時間を短縮します。スーパーコンピュータ「京」の多くの計算ノードと高速なノード間ネットワークを利用することで、短時間に大規模なメタゲノムデータを処理することが可能になりました。現在は、GHOSTMPを類似配列の検索手法として用いることで、微生物集団から得られた塩基配列に基づき系統解析及び機能解析までを高速に行う解析パイプラインの開発や、解析パイプラインを利用した実データの解析を進めています。
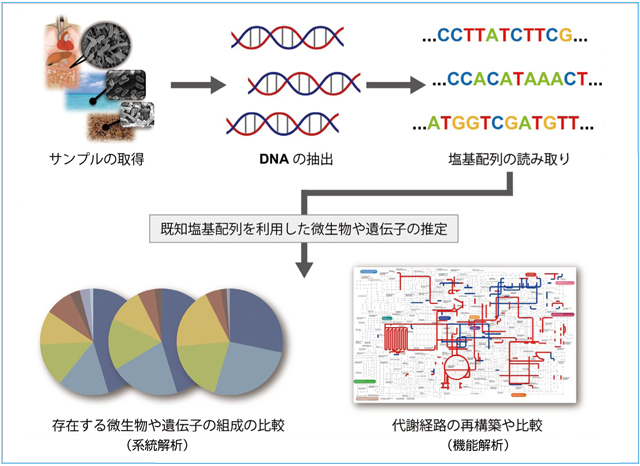
図1:メタゲノム解析の模式図

